テクノロジー
技術レポート:アーカイブ
Category:ライフサイエンス
PowerGeneデータベースの構築
ここ数年でDNAマイクロアレイを使った遺伝子発現解析は分子生物学研究において必須の実験手法となった。DNAマイクロアレイによる遺伝子発現データの蓄積が進むに伴い、発現データをコンピューター上で解析するプロセスもルーチン化されてきた。しかし、多数の遺伝子発現データだけから生物学的意味があるのかを突き止めることは非常に困難であり、研究者はゲノム、遺伝子、蛋白質、代謝といった世界中のデータベースを検索して、膨大な数にのぼる遺伝子プローブに関する知識を検索し、そこから意味があると思われるキーワードを抽出するという方法で遺伝子の機能を探り始めた。そのため、我々はこの課題において研究者を強力に支援する情報提供を行うことができる遺伝子発現プローブの翻訳/転写産物に関する知識を集約したデータベースPowerGeneを構築した。PowerGeneは当社製品BioINTEGRAの技術を使って構築されたデータベースであり、遺伝子発現プローブとそれに関する遺伝子のあらゆる情報を集約したWebサービスである。公共データベースは日々更新されるため、最新のデータを保つため自動アップデート機能を備え、Webインタフェースによる検索、保存、公開機能を実現した。最後にPowerGeneの情報を活用した知識抽出手段に関して考察し、自動アノテーションとマイニングプログラムにおける利用方法を示した。これは発現情報から遺伝子クラスタの機能を推定できるキーワードの抽出が可能な画期的な方法論である。
参考情報:
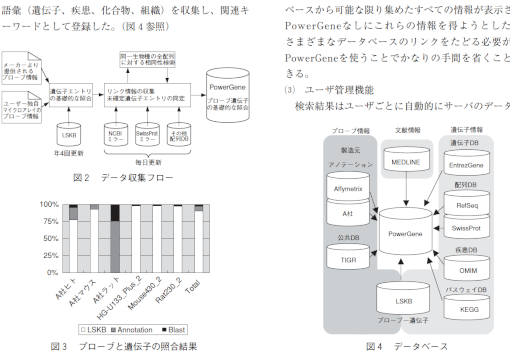
*関西事業部 バイオインフォマティクス部 MSS技報・Vol.18 39技術論文 PowerGeneデータベースの構築 PowerGene:a gene-based database linked directly to DNA Microarray probes松井 浩子* 谷嶋 成樹* 林 貴史* 上原 慶三* 石川 元一*Hiroko Matsui, Shigeki Tanishima, Takafumi Hayashi, Keizo Uehara, Motokazu Ishikawa ここ数年でDNAマイクロアレイを使った遺伝子発現解析は分子生物学研究において必須の実験手法となった。DNAマイクロアレイによる遺伝子発現データの蓄積が進むに伴い、発現データをコンピュータ上で解析するプロセスもルーチン化されてきた。しかし、多数の遺伝子発現データだけから生物学的意味があるのかを突き止めることは非常に困難であり、研究者はゲノム、遺伝子、蛋白質、代謝といった世界中のデータベースを検索して、膨大な数にのぼる遺伝子プローブに関する知識を検索し、そこから意味があると思われるキーワードを抽出するという方法で遺伝子の機能を探り始めた。そのため、我々はこの課題において研究者を強力に支援する情報提供を行うことができる遺伝子発現プローブの翻訳/転写産物に関する知識を集約したデータベースPowerGeneを構築した。PowerGeneは当社製品BioINTEGRAの技術を使って構築されたデータベースであり、遺伝子発現プローブとそれに関する遺伝子のあらゆる情報を集約したWebサービスである。公共データベースは日々更新されるため、最新のデータを保つため自動アップデート機能を備え、Webインタフェースによる検索、保存、公開機能を実現した。最後にPowerGeneの情報を活用した知識抽出手段に関して考察し、自動アノテーションとマイニングプログラムにおける利用方法を示した。これは発現情報から遺伝子クラスタの機能を推定できるキーワードの抽出が可能な画期的な方法論である。 Gene expression analysis using DNA microarray technology has been a fundamental part ofmolecular biology research, and then a basic process of computational analysis for expression datahas been being established. However, broad range of expertise of probes, about transcription andtranslation product, is necessary to find out what biological rules lie at the specific expressionpatterns. Therefore, most of researchers should search various databases such as genome, gene,protein, and pathway. In order to solve this problem we developed PowerGene database whichprovides various kinds of information about genes to which are linked probes. PowerGene databasealso provides latest information by automatic task, and can be access via the web interface.Additionally, we expanded PowerGene into an automated annotation function and a mining programas effective applications. 1.背景DNAマイクロアレイによる網羅的な遺伝子発現データを解析するため、様々なソフトウェアが開発されている(1)。これらソフトウェアのほとんどは、遺伝子発現値によるマイニングソフトウェアである。マイニングの内容は、遺伝子発現値を用いてクラスタリングやモデリングアルゴリズムにより遺伝子を分類し、実験条件との関連性を予測するものである。しかし、結局のところ遺伝子発現値によるマイニングソフトウェアでは遺伝発現クラスタ分類のレベルに留まっていた。遺伝子研究で本当に知りたい生化学的および生物学的意味の解明は、研究者自身の能力によるところが大きく、勘や経験による追加的実験で遺伝子の機能を解明してきた。遺伝子発現値によるマイニングソフトウェアの結果だけでは研究者のニーズと隔たりが大きかった。このような状況を少しでも改善するために、公開された配列アノテーション情報を用いて発現遺伝子の共通性を見つけることができるソフトウェアが開発されている(2)。しかし、遺伝子研究の成果は文献データベースに多くの情報が含まれており、配列アノテーションだけでは具体的な生化学的現象との関連性を結びつけることは難しい。我々のコンセプトは、遺伝子発現の解析に遺伝子配列と文献情報の両面からデータベース的アプローチを加え40技術論文ることで、エビデンスのしっかりした遺伝子マイニングを実現するというものである。規格化されて製品化されたDNAマイクロアレイを使用する場合には、アレイ製造元から提供されているデータベース(3)および多くのリンク情報を付加したデータベース(4,5,6)を参照することでプローブ遺伝子の情報を得ることができる。しかし、文献情報やそこから導かれる関連遺伝子や関連疾患の情報は少ない。また、規格外の生物種や独自のDNAマイクロアレイを使用する場合は、当然ながら情報検索が不可能となる。遺伝子研究の現場で求められる情報は、プローブの情報に限らず、プローブ遺伝子に関連するすべての情報であり、現時点では、世界中に分散した配列アノテーションや文献データベースに個別にアクセスして情報を集めるという方法しか無く、研究現場では情報収集作業の負担が増大している。遺伝子研究の現場で求められているデータベースに対する要求を整理すると、以下のようになる。①様々なメーカのDNAマイクロアレイのプローブと遺伝子のリンクが簡単に得られること②カスタムアレイ等、規格外のプローブや生物種に対応できること③遺伝子に関する世界中に散在した情報が集積されていること④文献情報を活用できること。特に、関連遺伝子、関連疾患、関連化合物等の重要なキーワードと遺伝子のリンクが取れていること⑤データベースのアップデートが容易なこと。遺伝子情報は日々更新され続けており、集められた遺伝子情報や文献情報は常に最新の情報を用いるべきである⑥遺伝子アノテーション、文献情報により、発現遺伝子のデータから遺伝子機能やアポトーシス等重要な生命現象との関連性をマイニングできること我々は、これらの研究現場ニーズを鑑みて、DNAマイクロアレイの種類に依存しないプローブ遺伝子データベースPowerGeneを開発した。すでに我々は公共データベースの自動アップデート機能、配列解析機能およびユーザ管理機能を備えた分子生物学研究支援ソフトウェアBioINTEGRAと、MEDLINE検索支援ソフトウェアMedRodeoの開発を完了しており、それらを応用することで最新の配列データベース、文献データベースを安定して活用できるデータベースとなっている。また、文献中の遺伝子表現を検出するためには、遺伝子名のシノニム辞書が必須となる。PowerGeneでは、ここに遺伝子および蛋白質辞書を搭載したワールドフュージョン社製LSKB(Life Science Knowledge Bank)を用いて高精度な検索を実現した。PowerGeneは創薬研究に必要なヒト、マウス、ラットのDNAマイクロアレイプローブと遺伝子を結びつけ、公共データベースから取得可能なできるかぎりのアノテーションを収集したデータベースである。LSKBをベースにプローブと遺伝子の関連と遺伝子に関するリンク情報を収集し、具体的なアノテーション情報をBioINTEGRA(配列アノテーション)とMedRodeo(文献データベース) を用いて収集した( 図1 )。PowerGeneを用いることでユーザは、すべてのプローブについて容易に最新情報を得ることができる。文献情報から得られたキーワードも充実しており、文献上での関連遺伝子、関連疾患、関連化合物、関連組織をキーワードとして登録している。また、規格外のDNAマイクロアレイに関しても、プローブの設計情報さえあれば配列アノテーションと文献データベースから自動的にプローブ遺伝子データベースを生成することができる。2.PowerGene2.1 データベース標準仕様のPowerGeneには、プローブエントリーとしてA f f y m e t r i x 製D N A マイクロアレイ( H G -U133_Plus_2、Mouse430_2、Rat230_2)が登録されている。また、ユーザーの手でAgilent製DNAマイクロアレイ(G4112A、G4122A、G4131A)やカスタムアレイのプローブを登録することが可能である。遺伝子はEntrezGeneに含まれるヒト、マウス、ラットのすべてのエントリーが登録されている。さらに、微生物の遺伝子や独自にシーケンスしたゲノムの遺伝子を登録することも可能である。アレイメーカーより提供されているプローブ情報には、遺伝子が未定義のまま残っているプローブも含まれている。配列データベースの基礎になるゲノムデータの更新が行われたときに発生する問題である。これらのプローブに対しては、最新のデータベースを照合することで遺伝子を探せる可能性がある。そこで、それぞれのプローブエントリーについて、毎日更新される公共データベースとの関連付けを行い可能な限りNCBI EntrezGeneとLSKB EntrezGeneAgilent/AffymetrixprobePowerGene図1 データ範囲MSS技報・Vol.18 41の関連性を追及した。(図2参照)このようにしてプローブとEntrezGeneエントリーとの関連付けの見直しを行った結果、平均して85%のプローブを遺伝子と関連付けることができた。この作業によりユーザーに提供されているプローブ情報の内容はアレイメーカーや製品によって様々であることも判明している。図3は、照合に成功した遺伝子に関して、プローブの照合結果を示すものである。LSKBには「A社ラット」を除き、メーカーから公開されたプローブ情報が登録されている。図3の”Annotation”で示されるプローブはメーカーが設計に用いた配列のデータベースエントリを用いて、EntrezGeneへのリンクを辿り直したものである。”Blast”で示したプローブは、遺伝子の核酸配列しか与えられていないものであり、最新の配列データベースに対して相同性検索をやり直したものである。次に、遺伝子についてより多くのアノテーションを収集するため、LSKB、BioINTEGRA、およびMedRodeoを用いて情報を抽出した。相同性検索により信頼性の高いリンク情報を有しているL S K B から、R e f S e q 、SwissProt、OMIMのアックセッションIDを取得し、GO、ドメイン、発現部位などのアノテーション情報をB i o I N T E G R A データベースから取得した。また、MedRodeoを用いて文献中で遺伝子と共出現頻度の高い語彙(遺伝子、疾患、化合物、組織)を収集し、関連キーワードとして登録した。(図4参照)2.2 ユーザインタフェースPowerGeneのユーザインタフェースはBioINTEGRAのフレームワークを使用している。BioINTEGRAの機能と組み合わせることで、検索にヒットした遺伝子の配列解析や文献解析、ユーザ管理を容易に行うことができる。盧クエリー入力画面検索キーワードは”Accession”,“Symbol”,“GeneName”,“Organism”,“Probe ID”,“Related Term”のいずれかを指定することができる。デフォルトでは全フィールドを検索する。“Related Term”フィールドにはMedRodeoで解析した遺伝子と関連する遺伝子、疾患、化合物、組織名が格納されている。例えば“cancer”[RelatedTerm]というクエリーで検索すると、“cancer”と関連の深い遺伝子を抽出することができる。また、キーワードをAND,OR,NOTの論理演算で組み合わせてクエリー文を作成することもできる(図5.a)。盪検索結果検索結果は図5.bのように一覧表示される。一覧にはシンボル、種、遺伝子名、プローブID、関連語、他データベースへのリンクが表示される。それぞれシンボルをクリックすると図5.cのようにその遺伝子の詳細な情報を見ることができる。この詳細画面には、公共データベースから可能な限り集めたすべての情報が表示される。PowerGeneなしにこれらの情報を得ようとした場合、さまざまなデータベースのリンクをたどる必要があり、PowerGeneを使うことでかなりの手間を省くことができる。蘯ユーザ管理機能検索結果はユーザごとに自動的にサーバのデータベーPowerGeneLSKBプローブ-遺伝子A社TIGR製造元アノテーションAffymetrix公共DBEntrezGene遺伝子DB配列DBSwissProtRefSeqOMIM疾患DBKEGGパスウェイDBMEDLINEプローブ情報 文献情報 遺伝子情報図4 データベース0%25%50%75%100%A社ヒトA社マウスA社ラットHG-U133_Plus_2Mouse430_2Rat230_2TotalLSKB Annotation Blast図3 プローブと遺伝子の照合結果LSKBメーカーより提供されるプローブ情報ユーザー独自マイクロアレイのプローブ情報遺伝子エントリの基礎的な照合NCBIミラーSwissProtミラーその他配列DB毎日更新年4回更新リンク情報の収集未確定遺伝子エントリの同定同一生物種の全配列に対する相同性検索PowerGeneプローブ遺伝子の基礎的な照合図2 データ収集フロー42技術論文スに保存され、後日参照することができる。またこれらの結果は指定したユーザグループに公開することもできる(図5.d)。3.考察例えば、30,000プローブが載ったDNAマイクロアレイを用いて実験を行った場合、抽出したプローブ遺伝子すべてについて最新の研究成果を熟知している研究者は皆無であろう。研究者は知らない遺伝子に関する発現情報は無視するか、DNAマイクロアレイ製造元から提供されているデータベース(3)を検索して知識を得てから状況を予測するしか選択肢は無い。通常は勘や経験で候補遺伝子のあたりを付けてその周辺の情報のみを調査し、その他の遺伝子に関する情報は捨てられていたのが実態である。そこで、PowerGeneを使って発現遺伝子群のマイニング方法に関して検討した。例として、関連する機能(共通キーワード)の抽出や関連疾患毎でのグルーピングを試みた。通常、DNAマイクロアレイによる遺伝子発現データは、GeneSpringやSpotfire等の統計解析ソフトウェアで遺伝子発現パターン毎にグルーピングされる。グルーピングされた遺伝子群(遺伝子クラスター)には通常十から数百の遺伝子が含まれ、遺伝子発現データ上それらの遺伝子は同様の振る舞いを示している。例えば、ガン細胞と正常細胞との対比において同様の発現パターンを示した遺伝子クラスターや特定の化合物に対する反応において同じパターンを示した遺伝子クラスターなどである。これらの遺伝子は同じ機能を担っていたり、同じ転写制御を受けていたり、同じ部位で発現していることが考えられる。遺伝子クラスターは図6に示すとおり、疾患、化合物(医薬品)および組織を結びつけるハブ(Hub)として作用すると言われており(8)、もし、遺伝子クラスターに関する既知の共通情報が見つかれば、化合物(医薬品)と副作用の関連等を予測することが可能になる。PowerGeneに含まれる情報で、遺伝子クラスターの共通情報抽出に関して有用性が高いと思われるものは以下の項目である。①文献から得られた関連遺伝子、関連疾患、関連化合物、関連組織②発現組織、臓器③細胞内プロセス④細胞内局在性⑤配列構造上の類似性今回の例では、上記の中で①、②、③の一部および⑤の一部の抽出プログラムを開発した。(図7参照)マイニングの結果、遺伝子クラスターに関して以下の情報を抽出することができる。①遺伝子クラスターに関連するすべての文献から得られた関連遺伝子、関連疾患、関連化合物、関連組織②上記結果において、関連因子ごとに分類された、さらに小さな遺伝子クラスター③関連度の強さを示すスコア、情報源となった文献数④共通するMeSHターム。MeSHタームにより共通する細胞内プロセスを抽出することができる。⑤上記MeSHタームのスコアには、観測された関連遺伝子の発現強度を加味している⑥配列構造上の共通点として共通するドメイン⑦配列データベース上に記載された発現組織入力された遺伝子クラスターはこれら①~⑦の結果により、細分類されそれぞれ関連する因子(キーワード)と結び付けられる。研究者の知識に頼らず網羅的に関連情報を入手でき、再分類された遺伝子クラスターの因子との関連性を参照することで候補遺伝子の絞込みが可能になる。遺伝子化合物 疾患組織図6 マイニングイメージ図a. 検索画面b. 検索結果画面d. 履歴画面c. 詳細画面図5 ユーザインタフェースMSS技報・Vol.18 434.まとめPowerGeneはDNAマイクロアレイ実験結果から生物学的ルールを見つける上で、重要な手助けとなる。また、遺伝子を中心に集められた豊富なアノテーションは、DNAマイクロアレイのみでなく、さまざまな分子生物学研究に応用が可能であろう。DNAマイクロアレイも進歩しており、ゲノム上の遺伝子部分だけでなく、すべてのエキソンの発現が観測できるExon Arrayや、ゲノム全領域の発現が観測できるTiling Arrayが実用段階に入っている。PowerGeneに含まれる情報はそれらの新型マイクロアレイの解析に関しても非常に有用であると考えられるため、対応を進めていく予定である。参考文献盧Silicon Genetics,Redwood City,CA盪Smid M,and Dorssers LC. GO-Mapper: functionalanalysis of gene expression data using theexpression level as a score to evaluate GeneOntology terms. Bioinformatics. 2004 Nov 1;20(16):2618-25蘯http://www.affymetrix.com/jp/analysis/index.affx盻Tsai J,Sultana R,Lee Y,Pertea G,KaramychevaS,Antonescu V,Cho J,Parvizi B,CheungF,Quackenbush J. RESOURCERER: a databasefor annotating and linking microarrayresources within and across species. Genome Biol.2001;2(11)眈Liefeld T,Reich M,Gould J,Zhang P,TamayoP,Mesirov JP. GeneCruiser: a web service for theannotation of microarray data. Bioinformatics. 2005Sep 15;21(18):3681-2.眇Draghici S,Sellamuthu S,Khatri P. Babel'stower revisited: a universal resource for crossreferencingacross annotation databases.Bioinformatics. 2006 Oct 26.眄Sherlock G,Hernandez-Boussard T,Kasarskis A,Binkley G,Matese JC,Dwight SS,Kaloper M,Weng S,Jin H,Ball CA,Eisen MB,SpellmanPT,Brown PO,Botstein D,Cherry JM. TheStanford Microarray Database. Nucleic Acids Res.2001 Jan 1;29(1):152-5.眩田中利男、西村有平、角田 宏、北岡義国、中 充子:薬理ゲノミクスとファルマインフォマティクス、日本臨牀、60巻1号(2002-1):39~50図7 マイニング結果画面例