テクノロジー
技術レポート:アーカイブ
Category:ライフサイエンス
臨床ゲノム情報統合データベースMGeNDの整備と公開
クリニカルシーケンスは、がんの領域において、2015年頃から日本でも実施され始め、2019年4月にはがんクリニカルシーケンスの保険適応が始まる見込みである。クリニカルシーケンスでは、検査で検出される遺伝子変異の病的判定が不可欠であるが、判定にはクリニカルシーケンスの検査の結果等が蓄積されたデータベースを用いている。日本でも国立研究開発法人日本医療研究開発機構(AMED:Japan Agency for Medical Research and Development)が主体となって、病院で蓄積されているクリニカルシーケンスの結果を臨床情報と合わせて統合する「臨床ゲノム情報統合データベースプロジェクト」が立ち上げられ、データベースとしてMGeND(Medical Genomics japan variant Database)が構築された。MGeNDでは、日本人特有の遺伝子変異だけでなく、有名疾患関連DBと連携し、キュレーション現場で活用可能なデータベースとなっている。本稿では、MGeNDのコンセプトから画面、応用事例について紹介する。
参考情報:
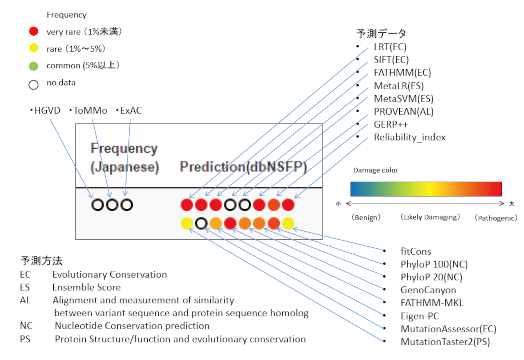
1 MSS 技報・Vol.29 臨床ゲノム情報統合データベースMGeNDの整備と公開 An integrated database of clinical and genomic infomation(MGeND) 野原 祥夫*Sachio Nohara クリニカルシーケンスは、がんの領域において、2015 年頃から日本でも実施され始め、2019 年4月にはがんクリニカルシーケンスの保険適応が始まる見込みである。クリニカルシーケンスでは、検査で検出される遺伝子変異の病的判定が不可欠であるが、判定にはクリニカルシーケンスの検査の結果等が蓄積されたデータベースを用いている。日本でも国立研究開発法人日本医療研究開発機構(AMED:Japan Agency for Medical Research and Development)が主体となって、病院で蓄積されているクリニカルシーケンスの結果を臨床情報と合わせて統合する「臨床ゲノム情報統合データベースプロジェクト」が立ち上げられ、データベースとしてMGeND(Medical Genomics japan variantDatabase)が構築された。MGeND では、日本人特有の遺伝子変異だけでなく、有名疾患関連DB と連携し、キュレーション現場で活用可能なデータベースとなっている。本稿では、MGeND のコンセプトから画面、応用事例について紹介する。 The clinical sequence of cancer has begun to be implemented in Japan from about 2015, andinsurance coverage is expected to begin in April 2019. In the clinical sequence, it is indispensableto judge the clinical significance of variants by using a database in which stored the results of clinicalsequence. In Japan, AMED( Japan Agency for Medical Research and Development) has started“clinical genome information integrated database project” which collects and integrates the clinicaland genomic information in the result of clinical sequence, and MGeND (Medical Genomics japanvariant Database) was opened with the non–restrictive access. MGeND is a database that can beutilized in curation, because it is integrated with the famous disease related DB and the Japanesevariant information. In this paper, we introduce the concept and application examples of MGeND. *関西事業部 バイオメディカルインフォマティクス開発室 1.まえがき 2018 年にがんゲノム医療中核拠点病院・がんゲノム医療連携病院の認定が行われ、国全体に「クリニカルシーケンス」が普及しつつある。「クリニカルシーケンス」では、検出された変異情報が病的な原因であるか否かを判定することが重要であり、これらの判断を行うには、疾患と変異情報を関連付けて管理されたデータベースが不可欠である。海外では、2006 年から開始されたTCGA プロジェクトを始め、Sanger Institute 社のCOSMIC(Catalogue OfSomatic Mutations In Cancer) ⑴、Memorial SloanKettering Cancer Center のOncoKB⑵等のデータベースが構築されている。その中でも米国国立衛生研究所が構築しているClinVar⑶は各病院・研究機関で検出された変異情報と臨床情報を組み合わせて登録、閲覧可能なデータベースであり、全世界的に変異の病的判定に使用されている。また、ClinVar ではClinGen ⑷と呼ばれるキュレーションWG と連携しており、ClinVar に登録されている変異情報のランク付けを行い、登録された変異情報のクオリティの担保を実現しようとしている。日本でも、慶應義塾大学 小崎 健次郎先生が構築されたDPV(Database of Pathogenic Variants)を始めとして特定の疾患をターゲットにした日本人疾患データベースが構築され、日本人特有の疾患変異情報の収集により、臨床・研究の現場で活用されている。その中で、AMED は2016 年に「臨床ゲノム情報統合データベースプロジェクト」⑸ を立ち上げ、研究課題「ゲノム医療を促進する臨床ゲノム情報知識基盤の構築」において各病院で実施されているクリニカルシーケンスにおいて検出された変異情報を疾患横断的に統合した。2018 年3月に臨床ゲノム情報統合データベース「MGeND」が非制限2 MSS 技報・Vol.29公開された。今回はMGeND とその使用方法について紹介する。2.MGeND本章では、MGeND のコンセプトとデータの受付について説明する。2.1 コンセプトMGeND は日本国内で蓄積されている疾患と遺伝子変異の情報を統合する目的で構築されている。MGeND に登録されるデータは、AMED が策定したデータシェアポリシーにのっとり、DS(Data Storage)(*♳ と呼ばれる医療機関から変異データが提供され、そのデータは制限公開又は非制限公開される(図1)。ゲノムデータは2017 年5月の個人情報改正法により、要配慮個人情報となり、取扱いには細心の注意を払う必要があるが、これらの問題を解決するために、国立研究開発法人国立国際医療研究センターにMGeND 登録管理委員会が設置され、提供された臨床・変異情報が非制限に公開できるか確認を行っている⑸。MGeND の特徴としては、入っているデータが日本人のみであり、様々な疾患と関連付けられて管理されていることである。登録されるデータが日本人のみであることから、ClinVar と比べてデータ数が少ない。臨床現場で使ってもらうためにMGeND では、この課題を「有名疾患関連DB との統合」、「予測データによる補完」の2つの方法で解決しようとしている。「有名疾患関連DB との統合」では、ClinVar を始めとする世界的に活用されているデータベース(表1:2018 年10 月時点21 個)と統合・比較することで日本人特有の変異情報を洗い出すとともに、MGeND に登録されていないデータの補完を実現する。「予測データによる補完」では、有名疾患関連DB にも登録されていない遺伝子変異における構造的変化をあらゆるコンピュータ予測ツールで算出した結果を一覧表示し、遺伝子変異の影響度の評価に活用する。MGeND は、蓄積された日本人変異情報だけでなく、有名疾患関連DB、予測データを組み合わせることで、「日本版ClinVar」と呼ばれ、「ゲノム医療における現場の研究者がキュレーションに使えるデータベース」、「ゲノム医療の実臨床においてカンファレンス時に参照できるデータベース」をコンセプトに構築されている。図1 MGeND のデータ公開までの流れ希少・難治がん感染症認知症難聴提供データ公共データData Storage (DS)定期的に更新MGeND(京都大学医学部付属病院)MGeND登録管理委員会(国立国際医療研究センター)倫理関係書類データ登録許可臨床・変異データ受付表1 有名疾患関連DBデータベース名バージョンEntrez Gene 20170908Gencode 28Disease Ontology 20180620ClinVar 2018–03COSMIC occurrence 85dbSNP b150CIViC 2018–03–01SnpEff –HGVD 2.3Human Phenotype Ontology 2018–07–25Clinical Trials 201706Insert disease from ICD10 –MedGen 20180516MeSH 20180711Orphanet V2.6ToMMo SNP 1ExAC 1MMMP 20160818DisGeNET 4.0Drug list 20160209GWAS catalog 201808293 MSS 技報・Vol.292.2 データ受付MGeND では、データ項目の性質に合わせて、4つのデータ項目でデータの受付を行っている。これは、各病院から提供されるデータを標準化することで、データの均一化、及び更新作業の負荷を軽減することが目的になっている。早速、データ形式について説明する。まずは、単一遺伝子疾患に関連するSNV、INDEL 等の変異データがあり、各患者単位での情報を格納した変異データと患者グループの中で該当の変異の出現頻度を計算した変異の頻度データが登録できる。がんや希少・難病における変異データが主に登録されている。次に、多因子遺伝子疾患に関連するSNV、INDEL 等の変異を格納したGWAS データがある。GWAS データでは、統計解析の結果、有意と判定(p–value < 0.05)された変異のみが格納されている。GWAS データでは、解析内容に合わせて解析単位を階層化して管理できるようになっており、感染症における解析データが主に登録されている。最後に、白血球の血液型として認識されているHLAのallele 情報がある。HLA の型によって、疾患への耐性等が理解でき、感染症における解析データが主に登録されている。これらの受付データに関する詳細な内容を表2にまとめる。受け付けたデータを有名疾患関連DB と関連付けて非制限で公開している。3.各画面の紹介MGeND のトップ画面を図2に示す。トップ画面から、①遺伝子名、変異名での検索、及び②疾患領域からの検索の2つの検索を用意している。また、キュレーション等に活用できるように、解析サービスで納品されたVCF 形式での検索も「③ Advanced Search」で実施できるようになっている。まずは検索した結果の一覧画面についてご紹介する(図3)。MGeND では、キュレーションに必要な情報を一括表示されるように設計されており、MGeND に登録されている日本人疾患変異データと共に、ファクトデータ(有名疾患関連DB の情報)とコンピュテーショナルデータ(予測データ)を統合表示している。その中でも予測データはあらゆる予測ツールで検出した結果であり、数字だけではどの程度構造的に影響があるのがわからないため、図4のように構造的影響度をグラデーションにて視覚的にわかるようにしている。個別の変異の詳細情報に関しては、MGeND における臨床統計情報と有名疾患関連DB のリファレンス情報を統合して表示し、変異のエビデンスの確認に活用可能である(図5)。リファレンス情報には各有名疾患関連DBへのリンクが用意されており、より詳細な情報にアクセスできるようになっている。疾患領域からの検索においては、疾患領域で登録された遺伝子、疾患の分布や、各疾患における登録統計情報等を表示している(図6)。また、各疾患特異的に取得されているデータも表示できるようになっており、認知症においては遺伝的リスクと関連付けられているAPOE遺伝型を表示しており⑹、感染症においては、HLA データの比較解析結果を表示している。これらのデータは、HLAの型をベースに疾患のリスクを判断する基盤となる。4.応用事例の紹介当社では、がんゲノムデータ解析サービスを全国の病院に展開している⑺。解析サービスでは、1症例当たり、数十件の変異が検出され、大半の症例で疾患の原因となる遺伝子異常が特定されているが、一部の症例では意義不明な変異しか検出されない場合がある。そのような場合、今後データが蓄積されることで、MGeND で日本人特有の疾患遺伝子変異でないか判定することができる。今回、クリニカルシーケンスを実施したときに、疾患遺伝子変異が見つからず、意義不明のBRCA2 I2149* の変異が検出されたと想定する。まず、トップ画面で「BRCA2 I2149*」で検索する(図7)。検索結果から、該当の変異が表示され、MGeND では、Pathogenic であるが、ClinVar を始めとした有名疾患関連DB では登録がないため、有名疾患関連DB だけでは変異の意義が特定できず、MGeND を使うことで変異の意義が特定できることになった。このような活用により、クリニカルシーケンスの検出精度向上が期待されることから、クリニカルシーケンスの普及において本データベースの重要性が向上すると考えられる。5.むすび2018 年3月に公開されてから、多くの変異情報が提供され、登録されている。また、疾患特異的な情報も提供され始め、疾患横断的なデータベースとして稼働し始め表2 データ項目データ項目登録データファイル形式変異データSNV、short INDEL、構造変異XML 形式、TSV 形式、XLSX 形式、VCF 形式変異の頻度データSNV、short INDEL、構造変異XML 形式、TSV 形式、XLSX 形式GWAS データHLA allele XLSX 形式HLA データ多型及び変異(SNV、 INDEL) XLSX 形式4 MSS 技報・Vol.29①遺伝子名、変異名等から検索②疾患領域からの検索③Advanced SearchVCF形式で入力可能図2 MGeND のトップ画面図3 変異一覧画面MGeNDに登録された日本人疾患変異データファクトデータ(有名疾患関連DBの情報)コンピュテーショナルデータ(予測データ)検索結果一覧から、疾患(疾患横断的)、薬剤、遺伝子情報でフィルタリングデフォルトではMGeNDに登録された変異のみ5 MSS 技報・Vol.29予測方法EC Evolutionary ConservationES Ensemble ScoreAL Alignment and measurement of similaritybetween variant sequence and protein sequence homologNC Nucleotide Conservation predictionPS Protein Structure/function and evolutionary conservation予測データ• LRT(EC)• SIFT(EC)• FATHMM(EC)• MetaLR(ES)• MetaSVM(ES)• PROVEAN(AL)• GERP++• Reliability_index• fitCons• PhyloP 100(NC)• PhyloP 20(NC)• GenoCanyon• FATHMM-MKL• Eigen-PC• MutationAssessor(EC)• MutationTaster2(PS)・HGVD ・ToMMo ・ExAC小大(Benign) (Likely Damaging) (Pathogenic)Frequencyvery rare (1%未満)rare (1%~5%)common (5%以上)no dataDamage color図4 変異の構造的影響度の表示変異のサマリ情報MGeNDにおける臨床統計情報有名疾患関連DBのリファレンス情報図5 変異の詳細情報6 MSS 技報・Vol.29疾患領域における登録の統計情報(遺伝子、疾患)疾患特異的なデータ表示(左:APOE遺伝子リスク、右:HLA比較データ)図6 疾患の統計情報有名疾患関連DBにはエビデンスがないMGeNDではPathogenicとして判定有りMGeNDに登録されている情報の統計情報が表示される図7 日本人特有の疾患変異7 MSS 技報・Vol.29ている。今後はよりクリニカルシーケンスの現場で活用可能な形でデータを登録、閲覧できる仕組みを実現し、より多くの日本人の変異情報が収集されていくと期待している。そのためには、海外の公共データベースと連携し、巨大化するデータベースへの対策(更新、速度)が必要であり、また、クリニカルシーケンスを行っている病院が変異・臨床情報を容易に登録できる仕組み作りが必要である。最後に、本プロジェクトを支援くださっている国立大学法人京都大学医学部奥野研究室の皆様には、深くお礼を申し上げる。本研究は、AMED の課題番号JP18kk0205013 の支援を受けて開発されている。 *1 DS とは、AMED の「臨床ゲノム情報統合データベースプロジェクト」において採択されたクリニカルシーケンスを実施している病院であり、11 の機関が代表機関として指定されている。参考文献(1) Forbes,S. A.,Beare,D.,Boutselakis,H.,et al.:COSMIC:somatic cancer genetics at high–resolution,Nucleic Acids Research,45,D1,D777~ D783(2016)Database URL:https://cancer.sanger.ac.uk/cosmic(2) Chakravarty,D.,Gao,J.,Phillips,S. M.,et al.:OncoKB:A Precision Oncology Knowledge Base,JCO Precision Oncology,2017,No.1(2017)Database URL:http://oncokb.org/(3) Landrum,M. J.,Lee,J. M.,Benson,M.,et al.:ClinVar:public archive of interpretations ofclinically relevant variants,Nucleic AcidsResearch,44,D1,D862 ~ D868(2015)Database URL:https://www.ncbi.nlm.nih.gov/clinvar/(4) Rehm,H. L.,Berg,J. S.,Brooks,L. D.,et al.:ClinGen - the Clinical Genome Resource,The NewEngland Journal of Medicine,372,No.23,2235 ~2242(2015)(5) 奥野 恭史,中津井 雅彦,鎌田 真由美:疾患レジストリーと知識データベース,日本医師会雑誌,147,No.7,1395 ~ 1399(2018)(6) Yajima,R.,Tokutake,T.,Koyama,A.,et al.:ApoE–isoform–dependent cellular uptake ofamyloid–β is mediated by lipoprotein receptorLR11/SorLA,Biochemical and BiophysicalResearch Communications,456,No.1,482 ~ 488(2015)(7) 毛利 涼,岡村 容伸,野原 祥夫,谷嶋 成樹:がんゲノムデータ解析:臨床現場への実装,MSS 技報,27(2017)http://www.mss.co.jp/technology/report/pdf/27_04.pdf執筆者紹介野原 祥夫1998 年入社。関西事業部へ配属。電力系統制御システムのソフトウエア開発に従事後、1999 年からバイオインフォマティクス・ゲノム解析のシステム開発に転向。2016 年から、AMED「臨床ゲノム情報統合データベース整備事業」における「ゲノム医療を促進する臨床ゲノム情報知識基盤の構築」の分担者として参加している。