テクノロジー
技術レポート:アーカイブ
Category:ライフサイエンス
創薬研究向け統合データウエアハウスシステム”TargetMine”の紹介
創薬研究のためのデータウエアハウスシステム“TargetMine”を紹介する。TargetMineは、世界の研究機関が公開している、遺伝子、タンパク質構造、疾患、医薬品等、創薬研究に関わる計23のデータベースを1つの統合されたオブジェクト指向データモデルに格納し、Webインターフェースによる簡便な操作により、情報の抽出、解析ができるシステムである。TargetMineはデータウエアハウスの考えに基づき構築されており、ベースとなった汎用データウエアハウス構築フレームワークInterMineについても併せて紹介する。現在、非商用目的であれば、TargetMineはhttp://www.mss.co.jp/businessfield/bioinformatics/solution/products/targetmine/index.htmlから自由に利用することができる。
参考情報:
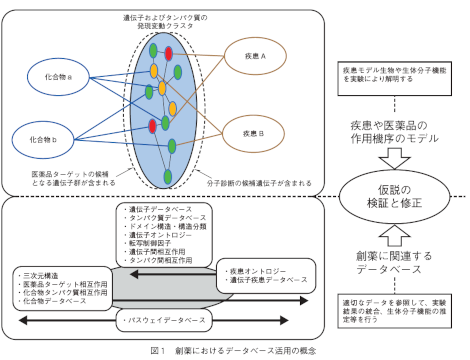
11 *関西事業部 第五技術部 創薬研究向け統合データウェアハウスシステム"TargetMine"の紹介 Introduction of TargetMine: data warehouse system for drug discovery 石川 元一* 谷嶋 成樹* Motokazu Ishikawa, Shigeki Tanishima 創薬研究のためのデータウェアハウスシステム“TargetMine”を紹介する。TargetMineは、世界の研究機関が公開している、遺伝子、タンパク質構造、疾患、医薬品等、創薬研究に関わる計23のデータベースを1つの統合されたオブジェクト指向データモデルに格納し、Webインターフェースによる簡便な操作により、情報の抽出、解析ができるシステムである。TargetMineはデータウェアハウスの考えに基づき構築されており、ベースとなった汎用データウェアハウス構築フレームワークInterMineについても併せて紹介する。現在、非商用目的であれば、TargetMineはhttp://targetmine.nibio.go.jp/から自由に利用することができる。 TargetMine is a data warehouse system for drug discovery. Data model of TargetMine is objectorientedand composed of 23 publicly available drug discovery related databases, includingdatabase of genes, protein structures, diseases, medical drugs, etc. Using well-designed webinterface, users can extract information and analyze data. InterMine, which is a framework forconstructing data warehouse system and was used for TargetMine, is also introduced. For noncommercialpurpose, TargetMine is freely available at http://targetmine.nibio.go.jp/ 1.はじめに 新規の医薬品を発見するためのプロセスである「創薬」には、化学構造、遺伝、タンパク質、疾患等の多種多様なデータが必要である。これらの各データベースは世界中の機関が提供している。例えばタンパク質立体構造情報を提供するProteinDataBank(1)である。近年の創薬研究においては、さまざまな形でデータベース化された知見に基づく仮説を立て、これらの各データを様々な切り口から統合的に分析し、情報を抽出し、その結果に基づいた仮説の立証、修正を要する。例えば、医薬品のターゲット遺伝子や分子診断の候補となる遺伝子の解明に関する仮説を立てる場合、図1に示すような作用機序モデルの概念に基づき対象となる生体分子の候補を求める。創薬に関連するデータベースはそれぞれ個別の現象に関する知見は表現されているものの、各機関が提供するそれぞれのデータベースを統合的に解析するための仕組みが欠けているという問題があった。この問題を解決するために医薬基盤研究所において開発されたのが、創薬研究向け統合データウェアハウス“TargetMine”(2)である。TargetMineにより、異なるデータベース間に共通する知見の抽出が容易となり、疾患や医薬品の作業機序の推定に必要な知識を網羅的に抽出することが可能になった。2.TargetMineのデータモデル 2012年11月現在、創薬に関する、表1の計23個のデータソースからのデータがTaregetMineに格納されている。対象の生物種は、創薬研究に直接関連する、ヒト、マウス、ラットが中心となっている。これらの各データベースのデータを統合するための、オブジェクト指向によるデータモデルが開発された。図2のデータモデルはこの一部である。TargetMineのWebインターフェースを用いることで、このデータモデルに沿った形で、データの検索、表示をすることができる。3.TargetMineのWebインターフェース TargetMineはWebアプリーケーションであり、トップページは図3のようになっている。3.1 クエリービルダー オブジェクト指向データモデルを構成する各クラスの一覧と、そのインスタンス数が図4のように表示される。オブジェクトの継承関係は階層的に表示される。例 MSS技報・Vol.23 12遺伝子およびタンパク質の発現変動クラスタ適切なデータを参照して、実験結果の統合、生体分子機能の推定等を行う疾患モデル生物や生体分子機能を実験により解明する疾患A疾患B化合物a化合物b医薬品ターゲットの候補となる遺伝子群が含まれる分子診断の候補遺伝子が含まれる・遺伝子データベース・タンパク質データベース・ドメイン構造・構造分類・遺伝子オントロジー・転写制御因子・遺伝子間相互作用・タンパク間相互作用・三次元構造・医薬品ターゲット相互作用・化合物タンパク質相互作用・化合物データベース・疾患オントロジー・遺伝子疾患データベース・パスウェイデータベース創薬に関連するデータベース仮説の検証と修正疾患や医薬品の作用機序のモデル図1 創薬におけるデータベース活用の概念カテゴリー種別名称文献InterMine共通タンパク質UniProtKB 3ドメインInterPro 4遺伝子間相互作用BioGRID 5遺伝子オントロジーGene Ontology、UniProtKB GOA 6遺伝子遺伝子Entrez Gene 7タンパク質(機能、立体構造)3次元構造PDBe SIFTS 8構造分類SCOP 9酵素The ENZYME database 10構造分類CATH 11タンパク質構造配列情報Gene3D 12疾患情報疾患オントロジーDisease Ontology 13疾患OMIM 14PPI、パスウェイPPI PPIview 15転写制御因子OregAnno 16転写制御因子Amadeus 17パスウェイNCI-Nature Pathway 18パスウェイReactome 19PPI iRefIndex 20パスウェイKEGG Pathway 21医薬品、医薬品-ターゲット情報医薬品ターゲット相互作用DrugBank 22化合物タンパク質相互作用STITCH 23化合物PubChem 24化合物ChEBI 25化合物ChEMBL 26表1 データベース一覧13に作用する医薬品リストを検索するクエリーを作成する場合を例にとって説明する。図5の左上の領域に、クラスと、それに関連するクラス、およびクラスの属性が表示されている。Compound Protein Interactionクラスは、医薬品とタンパク質との関連を表すクラスで、このクラスに、Assay、Compound、Data Set、Protein(タンパク質)の4つのクラスが関連づいていることが分かる。えば、図4では、Compound(化合物)のサブクラスとして、ChEBI Compound、ChEMBL Compound等の5つが存在することが分かる。この画面で、特定のクラスをクリックすることで、そのクラスを起点とした検索クエリーをクエリービルダー(図5)を用いて作成することができる。 ここで、代謝に関連する“cytochrome”タンパク質ProteinDomain- proteinDomain- primaryIdentifier:String 1- name:String- type:String- description:StringGene- gene 1 1 - interactWith11- symbol:String- name:String- ncbiGeneNumber:String- primaryIdentifier:String- secondaryIdentifier:StringProteinDNAInteraction- proteinDNAinteractions- interationType:String- name:String- role:StringProteinDNAExperiment- name:String- description:String- publication:PublicationBindingSite- bindingSite- sequence:String- start:Integer- end:Integer- chromosome:String- strand:String- genomeBuild:String- stableld:StringScopClassification- scopProtein:ScopProtein- scopSpecies:ScopSpecies- scopDomain:ScopDomain- scopFamily:ScopFamily- scopSuperfamily:ScopSuperfamily- scopFold:ScopFold- scopClass:ScopClass- scopClassification - scopRegions- scopRegions- chain- interactions- interactions- experimentProteinDomainRegion- proteinDomainRegions- targetProteins- proteinRegion1..*1..*1..*1..*1..*1..*1..*0..*0..*0..*0..*0..10..* - structureRelatedRegion- originalDb:String- originalId:String- start:Integer- end:IntegerDrug0..* 0..*0..*- drugBankld:String- description:String- genericName:String- brandNames:Synonym- drugTypes:DrugTypeChemicalCompound- compld:StringProteinStructure- pdbid:String- name:String- publication:PublicationProteinChain- pdbid:String- chain:String- identifier:String- organism:PdbOrganismScopRegion- start:Integer- startInsertionCode:String- end:Integer- endInsertionCode:String- chemicalCompounds- compounds- structures- structure 1DrugProteinInteraction- publications:PublicationPDBRegion- start:Integer- startInsertionCode:String- end:Integer- endInsertionCode:String- pdbRegions- pdbRegion- drugs0..*- drugs- drugProteinProteinStructureRegion1 - protein11 - protein11- protein11- chain- chains1- primaryldentifier:String- primaryAccession:String- name:String- length:Integer- molecularWeight:Integer- ecNumber:String図2 TargetMineのクラス図(Chen Y.(2011)(2)Fig.1を改変)図3 トップページ(TargetMine HPより引用)MSS技報・Vol.23 14また、Proteinクラスの属性がProteinの下に表示されていることが分かる。 各クラス、属性の右に表示されている“SHOW”ボタンをクリックすると、その情報を検索結果の表示として含めることを指定できる。各クラス、属性の右に表示されている“CONSTRAINT”ボタンをクリックすると、図6のようなダイアログが表示され、その属性に対する検索条件を入力することができる。“cytochrome”タンパク質を選びたい場合は、Proteinクラスのname属性のCONSTRAINTとして“cytochrome”を入力する。検索結果に表示する属性、および検索条件を指定すると、図5の右上の領域にその情報が表示される。ProteinのName属性に“CONTAINS cytochrome”という条件が表示されていることが分かる。 検索結果はテーブル形式に表示されるが、このテーブルのカラムの順序、およびソートするカラムを図5の下部で決定することができる。最後に、“Show Result”図5 クエリービルダー(TargetMine HPより引用)図4 クラス一覧(TargetMine HPより引用)15場合がある。例えば、発現解析により発現量が同時に増減することが判明した遺伝子群について、様々な情報を知りたい場合などである。このようなリストを検索条件に追加できるように、リスト機能がある。また、このリストについてエンリッチメント解析を簡単に行うことができる。図8の例は、ある遺伝子群に対する、パスウェイエンリッチメント、および化合物エンリッチメントの解析結果であり、パスウェイの結果を見るとAllograftrejectionのパスウェイがこのリストと最も強く関連づいていることがわかる。3.4 アカウント アカウント機能が用意されており、自分のユーザアカウントでログインすることで、クエリーや検索結果を保存しておくことができる。ボタンをクリックすると、検索結果が表示される。このように、クエリービルダーでは、GUIのみを用いて、情報検索を行うことができる。3.2 テンプレートクエリー このようにGUIを用いてクエリーをゼロベースで詳細に作成することも可能であるが、よく使用される検索については予め、テンプレートクエリーと呼ばれる検索のひな形が多数(2012年11月現在)用意されている。これらのうちの1つである「Gene(s)->All interactions」(図7)は、GeneのIDと生物種を入力することで、遺伝子と相互作用する遺伝子を簡単にリストアップすることができる。また、クエリービルダーでこれらのクエリーを基にしたクエリーを作成することもできる。3.3 リスト機能 複数のインスタンスを検索条件の制約として用いたい図6 検索条件の入力(TargetMine HPより引用)図7 テンプレートクエリー(TargetMine HPより引用)MSS技報・Vol.23 16InterMineでは、データモデルと、そのデータモデルのどこにそれぞれのデータソースからのデータを投入するのかをXMLを用いて記述する。汎用的なフォーマットにはパーサーが用意されているが、独自でパーサーを作成して用いることもできる。以上のものを準備することで、InterMineによるデータウェアハウスシステムの構築が可能であり、データベースへのデータ統合、Webインターフェースの作成も全てInterMineにより自動で実行される。 図9がInterMineのアーキテクチャの概要である。InterMineの内部構成は大きく3つに分類される。外部(ユーザー、スクリプト)とのインタラクションを行う部分(Web Application、Web Services)、リレーショナルデータベース(PostgreSQL)、そして、リレーショナルデータベースへの問い合わせを行い、ユーザー(スクリプト)から見えるオブジェクトベースのモデルと、3.5 API 以上の操作はWebブラウザからユーザーが実行するものであるが、これらを行うためのAPIも用意されている。Perl、Python、Ruby、JAVAの各言語向けのAPIが利用可能であり、これらの言語を用いてTargetMineの各機能を利用するようなプログラムを実装することが可能である。4.InterMine InterMine(http://www.intermine.org)(27)は、オブジェクト指向データウェアハウスシステムを構築するためのオープンソースソフトウェアであり、ケンブリッジ大学において開発されている。TargetMineはこのInterMineを用いて構築されており、他にもFlymine(28)やmodMine(29)などの他の生物学向けデータウェアハウスシステムの構築にもInterMineが用いられている。図8 エンリッチメント解析(TargetMine HPより引用)Object CacheWeb Services Object StoreWeb ApplicationAccess by scriptAccess by userSQLMaster TablePostgreSQLPrecomputedTableQueryOptimiserJAVA JDBCresult図9 InterMineのアーキテクチャ(Smith R.N( 2012) (26) Fig.1を改変)17用語解説・データウェアハウス 通常の業務に用いられる基幹系のシステムから得られたデータを投入したデータベースであり、情報分析、意思決定を行うために用いられる。データウェアハウスの概念を作ったWilliam H. Inmon氏の定義によれば、「経営者の意思決定を支援するための、目的別ごとに編成され、統合化された、更新処理を行わない時系列データの集まり」である。・創薬 創薬とは文字通り医薬品を創造するプロセスであり、医薬品となる化合物の機能の発見、また化学構造の設計、生物学的なアッセイなどの工程からなる。従来は、生体内はブラックボックスとして扱うよりなかったが、近年、疾患の分子的メカニズム、およびタンパク質構造の解明が急速に進んでいることから、バイオインフォマティクスを用いて創薬標的を同定し、その標的に対抗する化合物をゼロベースで設計するアプローチが多く用いられるようになっている。謝 辞 TargetMineの技術指導等でお世話になった医薬基盤研究所 水口 賢司 先生に感謝致します。参考文献⑴ Berman,H. M.et. al.(2000),'The Protein DataBank.',Nucleic Acids Res 28(1),235~242⑵ Chen,Y. et. al.(2011),'TargetMine,an integrateddata warehouse for candidate gene prioritisationand target discovery.',PLoS One 6(3),e17844.⑶ Apweiler,R. et. al.(2004),'UniProt:the UniversalProtein knowledgebase.',Nucleic Acids Res 32(Database issue),D115~D119⑷ Apweiler,R. et. al.(2000),'InterPro¦an integrateddocumentation resource for protein families,domains and functional sites.',Bioinformatics 16(12),1145~1150⑸ Stark,C. et. al.(2006),'BioGRID:a generalrepository for interaction datasets.',Nucleic AcidsRes 34(Database issue),D535~D539⑹ Ashburner,M. et. al.(2000),'Gene ontology:tool for the unification of biology. The GeneOntology Consortium.',Nat Genet 25(1),25~29⑺ Maglott,D. et. al.(2005),'Entrez Gene:genecenteredinformation at NCBI.',Nucleic Acids Resリレーショナルデータベース内のデータとの変換を行う部分(Object Store、Object Cache、Query Optimiser)である。 オブジェクトベースのクエリーは、Object StoreにおいてSQL文に変換されて、一旦Query Optimiserに渡される。データベース構築時に検索速度を高速化するため、Master Table以外にあらかじめ複数のテーブルを組み合わせたPrecomputed Tableが作成されている。Query Optimiserは、クエリーがPrecomputed Tableに合うものであればPrecomputed Tableを、なければMaster Tableを検索することで、検索速度を高速化している。検索結果はObject Storeに返され、ユーザーに渡すためにオブジェクトベースに変換されるが、後の検索に備えて検索結果の一部はObject Cacheにキャッシュされる。5.応用事例の紹介(疾患関連遺伝子機能アノテーション)と、GenomeJackとの連携について 変異ラットを用いた疾患関連遺伝子の探索において、TargetMineを用いて機能アノテーションを行った。疾患名をキーワードとする文献解析を用いて得られた特定疾患の関連遺伝子上位203個について、その遺伝子名をTargetMineに取り込み、ゲノム中の位置、Entrez GeneID、遺伝子シンボル、遺伝子機能情報をGenomeJackに取り込める形式で出力した。このデータをGenomeJackにインポートしてその後の解析を行った。既存の方法では事前準備、処理実行で2日間程度を要する作業であるが、TargetMineを用いることで試行錯誤の時間を含めても15分程度で上記データを出力できた。本事例により、TargetMineの優位性を再確認することができた。 また本事例により、高次機能のマイニング機能を持つTargetMineと、次世代ゲノムデータビューワーであるGenomeJackの連携の効果がとても大きいことが実証された。今後の開発によりTargetMineと、GenomeJackとの連携機能を強化することで、新規の知識情報の獲得プロセスを爆発的に加速させるプラットフォームを構築することが可能である(図10)。NGS 機器NGS データ解析実験計画へのフィードバックデータウェアハウス(TargetMine HP より引用)新規知見の発見図10 NGSパイプラインとの連携の構想MSS技報・Vol.23 18of Genes and Genomes.',Nucleic Acids Res 27(1),29~3422 Wishart,D. S. et. al.(2008),'DrugBank:a knowledgebasefor drugs,drug actions and drug targets.’,Nucleic Acids Res 36(Database issue),D901~D90623 Kuhn,M. et. al.(2008),'STITCH:interaction networksof chemicals and proteins.',Nucleic Acids Res 36(Database issue),D684~D68824 Wang,Y. et. al.(2009),'PubChem:a publicinformation system for analyzing bioactivities ofsmall molecules.',Nucleic Acids Res 37(WebServer issue),W623~W63325 De Matos P. et. al.(2009),‘Chemical Entities ofBiological Interest:an update.’,Nucleic AcidsRes 36(Database issue),D249~D25426 Gaulton A. et. al.(2012),‘ChEMBL:a largescalebioactivity database for drug discovery.’,Nucleic Acids Res 40(Database issue),D1100~D110727 Smith,R. N. et. al.(2012),'InterMine:a flexibledata warehouse system for the integration andanalysis of heterogeneous biological data.',Bioinformatics 28(23),3163~316528 Lyne,R. et. al.(2007),'FlyMine:an integrateddatabase for Drosophila and Anopheles genomics.',Genome Biol 8(7),R12929 Contrino,S. et. al.(2012),'modMine:flexibleaccess to modENCODE data.',Nucleic Acids Res40(Database issue),D1082~D108833(Database issue),D54~D58⑻ Velankar,S. et. al.(2005),'E-MSD:an integrateddata resource for bioinformatics.',Nucleic AcidsRes 33(Database issue),D262~D265⑼ Murzin,A. G. et. al.(1995),'SCOP:a structuralclassification of proteins database for theinvestigation of sequences and structures.',J MolBiol 247(4),536~540⑽ Bairoch,A.(2000),'The ENZYME database in2000.',Nucleic Acids Res 28(1),304~305⑾ Orengo,C. A. et. al.(1997),'CATH¦a hierarchicclassification of protein domain structures.',Structure 5(8),1093~1108⑿ Buchan,D. W. A. et. al.(2002),'Gene3D:structural assignment for whole genes andgenomes using the CATH domain structuredatabase.',Genome Res 12(3),503~514⒀ Osborne,J. D. et. al.(2009),'Annotating thehuman genome with Disease Ontology.',BMCGenomics 10 Suppl 1,S6⒁ Hamosh,A. et. al.(2005),'Online MendelianInheritance in Man(OMIM),a knowledgebase ofhuman genes and genetic disorders.',NucleicAcids Res 33(Database issue),D514~D517⒂ Yamasaki,C. et. al.(2010),'H-InvDB in 2009:extended database and data mining resources forhuman genes and transcripts.',Nucleic Acids Res38(Database issue),D626~D632⒃ Griffith,O. L. et. al.(2008),'ORegAnno:anopen-access community-driven resource forregulatory annotation.',Nucleic Acids Res 36(Database issue),D107~D113⒄ Linhart C. et. al.(2008),‘Transcription factorand microRNA motif discovery:the Amadeusplatform and a compendium of metazoan targetsets.’ Genome Res 18(7),1180~1189⒅ Schaefer,C. F. et. al.(2009),'PID:the PathwayInteraction Database.',Nucleic Acids Res 37(Database issue),D674~D679⒆ Joshi-Tope,G. et. al.(2005),'Reactome:aknowledgebase of biological pathways.',NucleicAcids Res 33(Database issue),D428~D432⒇ Razick,S. et. al.(2008),'iRefIndex:a consolidatedprotein interaction database with provenance.',BMC Bioinformatics 9,40521 Ogata,H. et. al.(1999),'KEGG:Kyoto Encyclopedia