テクノロジー
技術レポート:アーカイブ
Category:ライフサイエンス
次世代ゲノムブラウザー GenomeJackの開発
我々は、次世代ゲノムシーケンサに対応した全く新しいゲノムブラウザーGenomeJackを開発している。GenomeJackは、次世代シーケンサのマッピング済みリードデータおよび全ゲノムシーケンス、エクソーム、トランスクリプトームおよび変異解析に用いられるソフトウエアツールの結果を簡単な操作で閲覧することができるJavaアプリケーションである。GenomeJackは当社ダウンロードページ(http://genomejack.net/japanese/gj_download.html)よりダウンロードして誰でも無償で使用することができる。
参考情報:
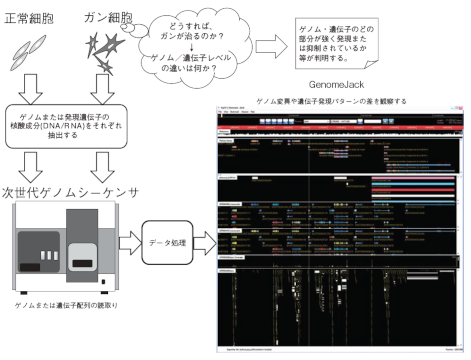
13 *関西事業部 第五技術部 次世代ゲノムブラウザ GenomeJackの開発 GenomeJack, The next-generation genome browser 谷嶋 成樹* 野原 祥夫* 石川 元一* 上原 慶三* 岡田 千尋* 小原 康雄* Shigeki Tanishima, Sachio Nohara, Motokazu Ishikawa, Keizo Uehara, Chihiro Okada, Yasuo Ohara 我々は、次世代ゲノムシーケンサに対応した全く新しいゲノムブラウザGenomeJackを開発している。GenomeJackは、次世代シーケンサのマッピング済みリードデータおよび全ゲノムシーケンス、エクソーム、トランスクリプトームおよび変異解析に用いられるソフトウェアツールの結果を簡単な操作で閲覧することができるJavaアプリケーションである。GenomeJackは当社ダウンロードページ(http://www.mss.co.jp/businessfield/bioinformatics/solution/products/genomejack/english.html)よりダウンロードして誰でも無償で使用することができる。 Recent 10 years, we developed several software products for genome-informatics data analysis.The GenomeJack is Java application program and it has been developed as novel genome-browsersoftware for a flood of genome-sequencing data as results of that many improvements of nextgenerationgenome sequencing platforms. GenomeJack provides quick operation and intuitive visualization "at a glance" for biologicalresearcher against such very large genome data sets and to flexibly integrate various data types,including not only sequencing data but also micro-array data. And visualization function ofGenomeJack is including genome-mapping data and resulting data from analysis software tools forwhole-genome and exome sequencing, epigenome and transcriptome analysis, and structuralvariants and copy number profiling. Now the GenomeJack is freely downloadable from an official web-site of MITSUBISHI SPACESOFTWARE(http://www.mss.co.jp/businessfield/bioinformatics/solution/products/genomejack/english.html). 1.まえがき 当社では、「千ドルゲノム解析」時代に向けて「次世代ゲノムブラウザ GenomeJack」を開発した。 近年の次世代ゲノムシーケンサの技術革新により数年以内に「千ドルゲノム解析」が実現し、創薬、医療および健康分野で革新的なゲノム情報の応用が始まると言われている。そして、「千ドルゲノム解析」の成否には、処理速度が速い解析ソフトウェアと使いやすいゲノムビューアが鍵を握っていると言われている。そこで当社では、本格的な「千ドルゲノム解析」の到来に備えて、研究者にとって軽快に動作し、使いやすい次世代ゲノムブラウザを製品化した。 西暦2000年前後に起こったヒトゲノム解読ブーム以降、当社ではゲノム機能解析に応用できる様々なソフトウェアを製品化してきており、それらは現在、様々な応用研究分野で活用されている。GenomeJackは、これまでの当社の製品開発ノウハウを活かして独自開発した次世代ゲノムブラウザである。 現在GenomeJackは無償ソフトウェアとして当社ウェブサイトからダウンロード形式で提供されており、簡単なアクティベーション操作により誰でも利用できるようになっている。2.次世代ゲノムシーケンス時代の到来2.1 次世代ゲノムシーケンサの特徴 2005年前後に次世代シーケンサが出現して以来、1塩基当たりの解析コストが年率約1/5以下の割合で低下しており、研究現場では急速に応用範囲が広がっている(図1)(1)(2)(3)。 次世代ゲノムシーケンサの特徴は、これまで主に用いられてきたサンガ―法のゲノムシーケンサに比べて、高 MSS技報・Vol.22 14スとDNAマイクロアレイ解析のデータマイニングに適したウェブサーバーシステムであり、研究組織全体に解析サービスを共有する方式で運用されている。これらの製品は、ヒトゲノム解読が完了した後の「ポストゲノム時代」に発売開始し、現在では主に創薬研究に活用されている(図1)。 次世代シーケンサは継続的に性能が改善されており、それに伴って研究現場での活用も指数関数的に増加している。図1の通り、これまで遺伝子研究に用いられてきた代表的な実験デバイスであるDNAマイクロアレイによる文献の増加率は減少している。一方で次世代シーケンサに関連した文献数は大きな伸び率で推移しており、今後伸びはさらに加速すると予測されている。 今回紹介するGenomeJackは次世代シーケンサを活用した遺伝子研究用に開発したソフトウェアである。次世代シーケンサで生み出されるデータは前述の通り非常に大量であり、「見るだけ」の動作においてもソフトウェ速、かつ、低コストでゲノム配列をシーケンシングできる点に尽きる。例えば、代表的な次世代シーケンサの1つであるillumina社のHiSeq2000システム(4)は、1ラン(1回のシーケンサの運転)当たり600Gb(b:ベース。核酸配列1塩基を意味する単位)の配列読取り能力を有している。2000年時点での代表的なサンガ―法ゲノムシーケンサ ABI3700システムと比較すると、ラン当たり約125万倍、時間当たり約13万倍の解読速度である。HiSeq2000システムには今後発展型の出現が予定されており、潜在的にはラン当たり1Tbの能力を発揮できると言われている。2.2 ゲノム分野における当社の既存製品 当社は、2003年に「バイオ研究環境構築支援システムBioINTEGRA」(5)を発売開始以来、siRNA配列設計システム(6)および創薬研究プラットフォームBioElephant(7)を製品化してきた。これらの製品は、ゲノムデータベー2000年2005年2010年$1$10$100$1k$10 k100万塩基当たりのコスト100万塩基当たりのコストPubMed登録文献数10100100010000100000次世代ゲノムシーケンサ出現Roche/454 GS20DNAマイクロアレイに関連したPubMed文献数次世代シーケンサに関連したPubMed文献数塩基当たりのシーケンシングコスト(年率1/5.23)【当社ゲノム解析ソフトウェア製品の推移】バイオ研究環境構築支援システムBioINTEGRAsiRNA配列設計システムsiSNIPER創薬研究プラットフォーム BioElephant次世代ゲノムブラウザ GenomeJack図1 次世代シーケンサのコスト、関連文献数の推移と当社製品THE SEQUENCE EXPLOSION, nature,Vol.464 1 April 2010、Next-generation sequencing: adjusting to data overload. Monya Baker, Nature Methods 7, 495 - 499 (2010) およびWhat would you do if you could sequence everything?, Avak Kahvejian, John Quackenbush & John F Thompson, Nature Biotechnology 26,1125 - 1133 (2008) よりデータを合成して作成15⑵ Integrative Genome Viewer(IGV) (http://www.broadinstitute.org/igv/)(9) 米国BROAD INSTITUTEが開発し、無償ソフトウェアとして公開しているゲノムブラウザである。世界的な知名度ではUCSC Genome Browserと双璧をなすものである。Javaアプリケーション形式で提供されているため、プレゼンテーションなどで良く用いられる。IGVは以下の様な特徴を持つ。 ・Javaアプリケーションである ・操作性が良い。スクロール等の操作を軽快に行える ・個人PC上のデータを表示できる。特に生データ(リードデータ)の表示が容易である3.2 既存ゲノムブラウザの問題点 UCSC Genome Browserには以下の様な問題点がある。 ・ウェブシステムであるため、スクロール、ズームなどの操作レスポンスが非常に遅い ・ユーザデータの検索ができない ・セキュリティ上の問題からデータをインターネット上のUCSCサイトにアップロードできない場合がある ・表示データのフィルタリングなど、候補データの絞り込みが不可能である ・ユーザ側で生データ(リードデータ)の公開サイトを設置する必要がある 一方、アプリケーション形式であるIntegrativeGenome Viewer( IGV)にも以下の様な問題点がある。 ・大量のリードデータ、特にトラック当たり1億リード以上のデータ量では、表示が非常に遅くなる ・ユーザデータをクライアント/サーバー形式で共有して参照することが非常に困難アの処理負荷は非常に重いものになる。GenomeJackはその点の対策に注力し、次世代シーケンサのデータを「見る」ツールとして、軽快な動作を実現させている。これにより、ユーザはソフトウェア操作レスポンス上のストレスを感じることなく、必要なデータの抽出を行うことができる。 従来の当社製品とは異なり、GenomeJackはアプリケーション形式であるが、GenomeJackのデータ管理技術には、BioINTEGRAにて開発された独自のゲノムデータベースエンジンの技術が継承されている。この技術により、今後想定される次世代シーケンサの改良に伴うデータ量の増大に対応できるように設計されている。3.GenomeJackの開発経緯3.1 世界のゲノムブラウザ状況 ゲノムブラウザは次世代シーケンサのデータ解析結果を閲覧するためのソフトウェアであり、実験結果の評価を行うためには必要不可欠なツールである。 現在、次世代シーケンサデータに対応した以下の様なソフトウェアがリリースされている。⑴ UCSC Genome Browser(http://genome.ucsc.edu )(8) 米国カリフォルニア大学サンタクルーズ校が開発し、ウェブサービスとして公開しているゲノムブラウザである。UCSC Genome Browserは世界で最も知られているゲノムブラウザの1つであり、以下の様な特徴を持つ。 ・ウェブサービスである ・アノテーション情報が充実している ・ユーザデータをアップロードして表示させることが可能 ・リファレンスゲノムや多様なアノテーション情報を公開している図2 UCSC Genome Browserの表示画面例 図3 Integrative Genome Viewer(IGV)ダウンロードページMSS技報・Vol.22 16Ver.1.3を当社ウェブサイトから無償ソフトウェアとして世界中の研究者向けに公開している(図4)。4.GenomeJackを用いた次世代シーケンサデータ解析4.1 ゲノム機能解析の概念(図5) 基礎医学や分子生物学では、異なる状態の2つ以上の生体サンプルの遺伝子発現等、分子生物学的差異を明確 ・ユーザデータの検索ができない ・データの抽出操作が複雑で使いにくい ・Excel等から出力されたデータをそのままインポートできない3.3 GenomeJackの開発コンセプト 当社は次世代シーケンサのデータ受託解析ビジネスを手掛けており、解析結果の確認にはUCSC GenomeBrowserやIGVを利用してきたが、前述の問題点に悩まされていた。特に、性能の向上した最新の次世代シーケンサのリードデータのブラウジングでは、使用に耐えられない程度の遅い操作レスポンスであった。 そのような経験に基づき、以下のコンセプトで新規にゲノムブラウザの開発を開始した。 ・操作レスポンスと表示レスポンスにおいて、ユーザにストレスを感じさせない程度の軽快な動作を実現すること ・様々な計算機プラットフォーム上で快適にプレゼンテーションができること ・後段の高次解析処理や実験に供する候補データを簡単に検索、絞り込み、抽出できること その結果、2010年10月にβ版のリリース開始し、2011年4月にVer.1.0を公開した。現在では、改良が進んだ1データ処理正常細胞 ガン細胞次世代ゲノムシーケンサゲノムまたは遺伝子配列の読取りGenomeJackゲノム変異や遺伝子発現パターンの差を観察するどうすれば、ガンが治るのか?↓ゲノム/遺伝子レベルの違いは何か?ゲノム・遺伝子のどの部分が強く発現または抑制されているか等が判明する。ゲノムまたは発現遺伝子の核酸成分(DNA/R NA)をそれぞれ抽出する図5 ゲノム機能解析の概念図4 GenomeJackダウンロードページ(10)http://www.mss.co.jp/businessfield/bioinformatics/solution/products/genomejack/17胞内で機能している場合があり、これに関しても次世代シーケンサで解明することが可能である。 エクソンの組合せ、アンチセンス鎖およびmiRNA等の領域は非常に多様であり、これら検出された領域を簡単かつ確実に閲覧するソフトウェアがGenomeJackである。 GenomeJackにより、ユーザは図5に示すゲノム上の遺伝子構造と対比しながら、各サンプルから得られた実験結果の比較が可能になる。4.2 次世代シーケンサによるゲノム機能解析の原理(図7) サンプル細胞から抽出されたDNAまたはRNA分子はシーケンシングの前処理段階で数100b~数kbの長さで断片化され、次世代シーケンサにより塩基配列を読み取られる。塩基配列の読取り長はシーケンサの種類や運転条件によって異なるが、100b程度の場合が多い。ランで600Gbの読取りができるHiSeq2000の場合は、100bのリード配列が約60億個出力される。 読み取られたリード配列はゲノム配列の一部であるため、既知のゲノム配列との照合により染色体中の場所が判明する。これをマッピングという。マッピングは既知ゲノム配列とギャップやミスマッチを考慮した最適アライメントにより行われる。 一般に、次世代シーケンサにより読み取られる核酸配列の数は、リファレンス配列の塩基数の10倍~30倍程度の密度で読み取られる。これを冗長度10~30という。また、局所的な冗長度のことをカバレージという。カバレ化することにより疾患の原因遺伝子や化合物の作用機序を解明する。そのような場合に、GenomeJackはサンプル間のDNA/RNAの変動、発現の差を明確に表示するために用いられる。例えば図5に示す通り、特定のガンの治療法を研究する場合、正常細胞とがん細胞の遺伝子発現パターンの差から原因遺伝子を特定することがある。次世代シーケンサにより、正常細胞およびガン細胞内に存在しているRNA分子の分布と量を測り、その差を求めて、大きな差異のあった部分の近傍に存在する遺伝子を疾患の原因遺伝子の候補として抽出する。従来のRNAマイクロアレイを用いた遺伝子発現解析法では、あらかじめ決められた遺伝子領域の一部分のみをプローブとして利用するため、遺伝子領域全体の状態を解明することはできなかった。 ゲノムDNA上の遺伝子の構造は図6に示す通りである。ゲノム上の遺伝子コーディングリージョンには「エクソン」および「イントロン」と呼ばれる領域があり、スプライシングにより「エクソン」領域だけが連結されてmRNA(メッセンジャーRNA)となり、タンパク質に翻訳されて生体内で機能する。次世代シーケンサによる解析では、エクソン単位での解析が可能になるため、スプライシングバリアントを含むすべての遺伝子発現パターンを解明できる。一部のイントロンに相当する配列に対して「アンチセンス鎖」と呼ばれる遺伝子発現を抑制する分子が存在する場合があり、これも次世代シーケンサで解析可能である。また、遺伝子と遺伝子の間の領域からmiRNAと呼ばれる短いRNA分子が翻訳されて細プロモーターエクソンゲノムDNAスプライシングメッセンジャーRNA(mRNA)アンチセンス鎖miRNA翻訳(タンパク質合成)次世代シーケンサによる解析では、これらをすべて解明することが出来るコーディングリージョン(CDS)様々な組合せ(スプライシングバリアント)を持つイントロン図6 遺伝子の構造MSS技報・Vol.22 18 スループットはラン当たり0.5~2Gbであり次世代シーケンサの中では少ない方であるが、有効リード長が400bと比較的長いことから、de-novo genome assemblyやde-novo transcriptome assemblyに適したシーケンサである。GenomeJackでは、de-novo genome assembly後のアノテーション表示、de-novo transcriptomeassemblyの結果をBLASTまたはBLATで既存ゲノム情報にマッピングした結果を表示できる。⑵ illumina Genome Analyzer IIx/HiSeq2000 (GAIIx/HiSeq2000) 有効リード長は50~150bpと短いが、スループットがラン当たり100Gb~600Gbと非常に大きく、次世代シーケンサの本命と言えるタイプである。変異解析、発現解析、エピゲノム解析等応用範囲は広い。スループット向上による塩基当たりの読取りコストの低減が進んだため、DNAマイクロアレイに代わる定量性の高い発現解析デバイスとして利用が進みつつある。 GenomeJackでは、以下のような解析ツールの結果を表示できる。 ・マッピングツール BAM/SAM形式に対応したマッパー。例えばbowtie(13)、BWA(14) ・ゲノムの多型解析ツール SAMtools(15) ・RNA-seq解析ツール tophat(16)、cufflinks(17)、cuffdiff(17)ージが2以上の場合はゲノム配列が重複して読み取られるため、カバレージが10以上の領域ではリード配列に0.1%程度含まれる読み取りエラーの判別が可能になる。 リファレンスゲノム配列に対するゲノム変異を解析する場合、すべての染色体上で均一に重複してリード配列が読み取られるようにサンプルが調製される。ゲノム上のすべての領域で均一なカバレージが保たれることにより、ヒトの個人差や特定作物の株に生じているゲノム変異を網羅的に解明することができる。 また、ゲノム上の特定の場所に結合しているタンパク質の分布を解明する場合は、タンパク質が結合している状態でゲノムDNAを断片化して、その断片を読み取ることにより、タンパク質が結合しているゲノム上の位置を特定することが可能になる(ChIP-seq法(11))。 ゲノムから発現している遺伝子のRNAを読み取った場合、RNA配列をゲノム上にマッピングすることにより、全遺伝子の発現強度を網羅的に解明することができる(RNA-seq法(11))。 なお、ChIP-seq解析やRNA-seq解析においても、カバレージが高い領域に関してはサンプルのゲノム変異(核酸種類の変異)が検出される場合が多い。4.3 GenomeJackが想定する次世代ゲノムシーケンサ GenomeJackでは以下のような次世代シーケンサのデータ表示を想定している。⑴ Roche Genome Sequencer FLX(GS FLX)(12)次世代シーケンサリファレンスゲノム配列へのマッピングリード配列サンプル細胞DNA/RNA抽出断片化断片化された核酸配列の解読(1ランで数億リード以上)リファレンスゲノム配列マップされたリード配列群既知遺伝子のポジション核酸種類の変異検出されたシグナル(遺伝子発現制御領域)ターゲットと考えられる遺伝子領域図7 ゲノム機能解析の原理(ChIP-seq解析の例)19販売されているが(20)、データ解析においてはGAII/HiSeq2000とほぼ同じソフトウェアが利用できる。4.4 GenomeJackによるデータ解析の流れ 図8にGenomeJackを使ったデータ解析の流れを示す。 GenomeJackは、解析パイプラインの各段階で発生するデータの閲覧が可能である。 一般に、解析対象となるサンプルデータは、正常細胞から得られたコントロールデータと実際に解析対象になるターゲットサンプルデータが何組も発生する。GenomeJackではそれらの解析データを、階層的に管理することができる(図9)。例えば、1つのコントロールサンプルに対して、時間経過毎のデータや、病態のステージ毎の細胞から得られたデータを、図9に示す階層構造にて管理することができる。 よって、ユーザはGenomeJackを解析結果の管理場所として活用することができ、必要に応じて検索や絞り込みを行った結果を、次の処理である三次解析、実験系や ・ChIP-seq解析ツール MACS(18) 上記以外でも、大規模変異解析、CNV(Copy NumberVariation)解析およびFusion Gene解析など、最新の解析ツールの結果やExcel等のスプレッドシートでユーザが下降してデータをTSV(タブ区切りテキスト)形式に変換してインポートし表示できる。⑶ ABI SOLiD 4(SOLiD4)(19) 次世代シーケンサのもう一つの雄である。GAIIx/HiSeq2000に近いスループットを有し、同様に有効リード長も50~75bpと短めである。color spaceと呼ばれる独自の塩基読取りフォーマットでデータが出力されるため、使用できる解析ソフトウェアに制限があったが、2009年前後からデータ解析に用いるフリーソフトウェアの対応が急速に進み、現在では問題なく使用することができる。使用できるソフトウェは、GAIIx/HiSeq2000と同様である。⑷ その他 Helicos社からもタイプの異なる次世代シーケンサがアノテーションリファレンスゲノムサンプル(1)リード・・・・・・コントロールサンプルターゲットサンプルマッピングQC解析サンプル間比較サンプル(2)リードマッピングQC解析公共DBよりリストSAM,BAMbed,bedGraphWiggle,GTF,TSVTSVFASTA,GFF,GTF,TSV,MySQL【一次解析ツール例】FASTQCbowtiebwa【二次解析ツール例】[Variant Analysis]samtools pileup[RNA-seq]tophat, cufflinks[ChIP-seq]MACS[サンプル間比較]cuffdiffExcel比較対照サンプル群のシーケンスデータインポートGenomeJack 解析パイプラインG enomeJ ackの機能を活用してゲノム上の候補領域を絞り込む【エクスポート】・候補領域リスト・配列データ・画像【三次解析へ】・パスウェイ解析・機能解析【実験系へ】・遺伝子クローニング・サンプル条件変更【論文作成へ】・画像データ・リスト・配列ユーザ図8 GenomeJackを使ったデータ処理の流れMSS技報・Vol.22 20論文作成に必要なデータ抽出に利用することができる。 なお、GenomeJackを使ったデータ解析の原理やコマンド等の詳細手順に関しては、参考文献(21)を参照されたい。5.GenomeJackのソフトウェア構造5.1 GenomeJackの内部構造(図10) GenomeJackはJava 1.6上で開発されたアプリケーションソフトウェアであり、Windows XP/Vista/7、MacOS XおよびLinuxベースのオペレーティングシステムでの動作を前提としている。 ビューは、解析データを表示するモジュールであり、トラックビューとテーブルビューから構成されている。ビューに表示するデータは、データインポータによりGenomeJackの内部形式に変換されてローカルファイルシステムに保存される。モデル・コントローラは、ローカルファイルシステムに保存されたデータに適宜アクセローカルファイルシステムモデル・コントローラ[gj]トラック表示設定トラック表示データ検索用インデックス[images]表示用のイメージ部品[lib]GenomeJack JavaライブラリプラグインJavaライブラリ[Settings]TSV、GTFファイルインポータ定義ファイル[Templates]トラック表示フォーマットファイル外部コマンド受信/外部サーバー参照データインポーター・エクスポータービュー・トラックビュー・ールーJava 1.6以上WindowsXP(SP2), Vista, 7Linux kernel 2.6以上Mac OS X 10.5以上染色体および表示位置切替コマンドhttpまたはhttpsG enomeJ ack S ervletリモートファイルシステムhttpリファレンスゲノム配列解析データ・GTF・bed / bedGraph・Wiggle・タブ区切りテキスト・マイクロアレイアノテーションデータ・GTF / GFFファイル・UCSC / Ensembl MySQLトラックイメージ・SVG・PNGトラックデータ・Fasta・bed・タブ区切りテキスト検索・絞り込み・ソートテーブルデータ・コンマ区切りテキストテブビュ図10 GenomeJackの内部構造図9 GenomeJackの登録データ管理画面(トラック管理画面)21る。GenomeJack Servletにより、ネットワーク上に配置された表示データを複数のクライアントから同時にアクセスすることが可能である。GenomeJack Servletを研究所内のイントラネット上に配置し、所内で解析データを共有することが可能になる。スし、トラックビューとテーブルビューに表示する。メモリ使用量が少なく、かつ、高速にデータにアクセスできるようにデータのブロック化が行われているため、32bitオペレーティングシステム上でも支障なく動作させることができる。また、データインポート時に、テーブルビューのデータに対してインデックスが生成され、高速な検索、絞り込み、ソートが可能になっている。 外部インタフェースとデータエクスポーターはGenomeJackの特徴的な部分である。従来のゲノムビューワは「見るだけ」の機能しか備わっていないものが多かったが、GenomeJackでは解析結果の利用を容易にするために、データエクポーターが充実している。トラックビューで見えている範囲のデータを、トラックイメージおよびトラックデータとして出力する機能、テーブルデータを一括で出力する機能が備わっている。 GenomeJackはhttpプロトコルにより、表示させる染色体と表示位置を変更することができる。この機能により、プレゼンテーションツール、スプレッドシートやウェブページからhttpのリンクによりGenomeJack画面を操作することができる。 また、GenomeJackはクライアント/サーバーシステムとして動作させることも可能である。これには、GenomeJack Servletというモジュールが用意されていtopfeature/subfeature表示(アノテーション表示)(スコアによる色変化あり)領域表示topfeature/subfeature表示(スコアによる色変化あり)設定ダイアログの呼び出し 一覧表の呼び出し図12 featureトラック表示例コンテナ フレームトラックメニュースケールコントローラゲージメッセージエリア(トラック表示例)リファレンスゲノムアノテーションサンプル間の差サンプル1発現パターンサンプル2発現パターンサンプル1カバレージサンプル1リード図11 GenomeJack表示画面の構造MSS技報・Vol.22 22ラックである。GenomeJackにおいてこれらの領域はfeatureと呼ばれており、図12に示す通り、単純に領域を表示する形式と、親子関係を持ったtopfeature/subfeature表示形式が利用できる。一般的にfeatureには、スコアやエラー確率などの数値が付加されており、これら数値をつかった表示色変化機能やラベリング機能を実現している。 featureトラックで表示されるデータは、同時にテーブルビューで一覧表形式にて表示することができる。一覧表上でデータを絞り込むことができ、同じ条件に従って、featureトラックに表示されている要素も絞り込まれる。5.4 グラフトラック(図13) 検出したリードのカバレージやシグナル値などの連続値を表示することができる。5.2 表示画面の構造 図11に示す通り、GenomeJackでは解析結果は「トラック」と呼ばれる領域に表示される。 各トラックの位置は、マウスのドラッグ&ドロップ操作により自由に入れ替え可能である。また、横方向のスクロールは、マウスのドラッグ、矢印キー操作、コントローラ上の操作ボタンにより可能である。横方向のスケールは、染色体全域表示から塩基種別が表示可能なレベルまで自由に拡大縮小が可能である。拡大縮小はマウスのダブルクリックとショートカットキーにより軽快に操作できるように考慮されている。5.3 featureトラック(図12) ゲノム上の遺伝子の位置を示すアノテーション情報や、解析の結果検出されたゲノム上の領域を表示するト図13 グラフトラック図14 リードトラック23できる。 ・クライアント/サーバーシステムを容易に構成できる 研究所内のイントラネット等で解析データを共有して閲覧できる環境を容易に構築できる。 当社はGenomeJackを通じてエンドユーザとの情報交換を密にし、次世代シーケンサによるゲノム解析技術の発展に寄与していきたいと考えている。GenomeJackに関しては、当社ウェブページの他に、Twitterによる情報発信も行っている。Twitterアカウントをお持ちの方は、是非genomejackをフォローしていただきたい。 今後、GenomeJackは世界のゲノムブラウザのデファクトスタンダードを目指して改良を加えながら、無償ソフトウェアとして提供を続ける予定である。次世代シーケンサでの解析を実践している研究者の方々には、是非軽快な操作感を味わってほしい。また、これから次世代シーケンサによる解析に挑戦していく方のために、データ解析のデモンストレーションデータ付きDVDの配布も行っている。詳しくは、GenomeJackのダウンロードページを参照されたい。参考文献⑴ THE SEQUENCE EXPLOSION, nature,Vol.464 1April 2010⑵ Next-generation sequencing: adjusting to dataoverload. Monya Baker, Nature Methods 7, 495~499(2010)⑶ What would you do if you could sequenceeverything?, Avak Kahvejian, John Quackenbush& John F Thompson, Nature Biotechnology 26, 図9に示す通り、透過性をもったグラフ表示機能が備わっているため、一つのグラフトラックに複数のグラフデータを重畳することが可能である。5.5 リードトラック(図14) 次世代シーケンサが読み取った塩基配列であるリードを表示するトラックである。リードの方向性、変異した塩基、リードのペア、ギャップの表現が可能である。6.むすび 従来のゲノムブラウザと比較した場合のGenomeJackの特徴は、以下のようにまとめることができる。 ・軽快な操作性 32bitオペレーティングシステムでも軽快にリードデータを閲覧できる。さらに64bitオペレーティングしシステムに移行すれば、将来予想される次世代シーケンサのスループット向上にも余裕を持って対応できる。 ・多様な入力データフォーマットに対応 業界標準のデータフォーマットに加えて、マイクロアレイデータファイル、任意のタブ区切りテキスト形式に対応できる。 ・データ出力機能が豊富 データを検索、絞り込み、ソートし、多様な形式で出力することができる。(FASTA、bed、CSV、PNG、SVG) ・多様なトラック形式、一覧表形式の表示 ・データ管理機能を有する 階層構造で表示データを管理できる。プレゼンテーション等で表示データを簡単に切り替えることが表1 入出力データフォーマットトラック種類用途インポート形式エクスポート形式リファレンストラックリファレンスゲノム表示FASTA FASTA画像データ(PNGまたはSVG形式)リードトラックマッピング結果表示SAMBAM -featureトラック変異解析結果表示VCF FASTA(IUPAC)アノテーション表示解析結果表示GFFGTFbedbedGraphMySQL(UCSC) タブ区切りテキストまたはbedシンテニー表示MFAMySQL(BioMart)マイクロアレイ表示ユーザデータ表示タブ区切りテキストグラフトラックカバレージ表示SAM/BAMWiggle(wig)検出シグナル強度表示Wiggle(wig)テーブルビューfeatureトラック要素の一覧表示featureトラックと同じCSVMSS技報・Vol.22 241125~1133(2008)⑷ http://www.illuminakk.co.jp/product/system/hiseq2000.shtml⑸ BioINTEGRA:http://www.mss.co.jp/businessfield/bioinformatics/solution/products/biointegra/index.html⑹ siSNIPER:http://www.mss.co.jp/businessfield/bioinformatics/solution/products/sisniper/index.html⑺ BioElephant:http://www.mss.co.jp/businessfield/bioinformatics/solution/products/bioelephant/index.html⑻ http://genome.ucsc.edu⑼ http://www.broadinstitute.org/igv/⑽ http://www.mss.co.jp/businessfield/bioinformatics/solution/products/genomejack/⑾ Shirley P. et.al.: Computation for ChIP-seq andRNA-seq studies.Nature Methods 6, S22~S32(2009)⑿ http://454.com/products-solutions/product-list.asp⒀ Ben L. et.al.: Ultrafast and memory-efficientalignment of short DNA sequences to the humangenome. Genome Biology 2009, 10:R25⒁ Li H. et.al. :Fast and accurate long-read alignmentwith Burrows-Wheeler Transform. Bioinformatics,Epub(2010).⒂ SAMtools :http://samtools.sourceforge.net/⒃ Trapnell C. et.al.:TopHat: discovering splicejunctions with RNA-Seq. Bioinformatics. 2009 May1; 25(9):1105~1111⒄ [12] Trapnell C. et.al. :Transcript assembly andquantification by RNA-Seq reveals unannotatedtranscripts and isoform switching during celldifferentiation. Nature Biotechnology NatureBiotechnology 28, 511~515( 2010)⒅ [10] Zhang et.al. :Model-based Analysis of ChIP-Seq(MACS). Genome Biol(2008)vol. 9(9)pp. R137⒆ http://www.appliedbiosystems.jp/website/jp/product/modelpage.jsp?BUCD=121824&PLCD=121823&MODELCD=121822⒇ http://www.helicosbio.com/Products/Helicosreg-G e n e t i c A n a l y s i s S y s t e m / H e l i S c o p e t r a d e -Sequencer/tabid/87/Default.aspx21 谷嶋 成樹・石川 元一:ゲノムインフォマティクス-次世代シーケンサデータ解析の実践、最新創薬インフォマティクス活用マニュアル 創薬研究シリーズ/遺伝子医学MOOK別冊 2011 メディカルドゥ